Bakteriofag, foto: DTUBakteriofager er virus, der angriber bakterier. I den proces er de blandt andet med til at påvirke bakteriers udvikling og deres evne til at gøre mennesker og dyr syge samt overføre antibiotikaresistens. Viden om bakteriofager giver os større indsigt i livets evolution og kan føre til udvikling af bedre medicin og værktøjer til at ændre på gener.
Datasættet inkluderer danske prøver
Et internationalt forskerhold anført af University of California, Berkeley, og med deltagelse fra DTU Fødevareinstituttet har opdaget mange nye slags kæmpe-bakteriofager ved at undersøge ukendt DNA fra bl.a. dyre- og menneskeafføring, vand og jord indsamlet fra hele kloden.
De har derved fundet og rekonstrueret arvematerialet fra de største bakteriofager, verden endnu har set. Eksisterende viden baseret på traditionelle analysemetoder har indikeret, at bakteriofag-genomer er små.
Men ved at bruge nye metoder er det lykkedes forskerne af finde en lang række kæmpe-bakteriofager og dermed en større diversitet end hidtil set, ligesom forskerne har genereret ny viden om kendte bakteriofager. Faktisk har de afdækket hele genomet for ikke mindre end i alt 175 kæmpe-bakteriofager. Det vil sige, at hvert eneste nukleotid i deres DNA er kortlagt.
Selvom fager og andre virus per definition ikke er levende, bærer de nyopdagede genomer rundt på mange gener forbundet med stofskifte og immunforsvar, hvilket udvisker grænsen for, hvad vi betragter som cellulært liv og simple molekylære parasitter.
Postdoc Patrick Munk ved DTU Fødevareinstituttet har bidraget til studiet ved at identificere, samle og genopbygge store bakteriofag-genomer fundet i dansk svineafføring.
Navne henviser til forskernes nationalitet
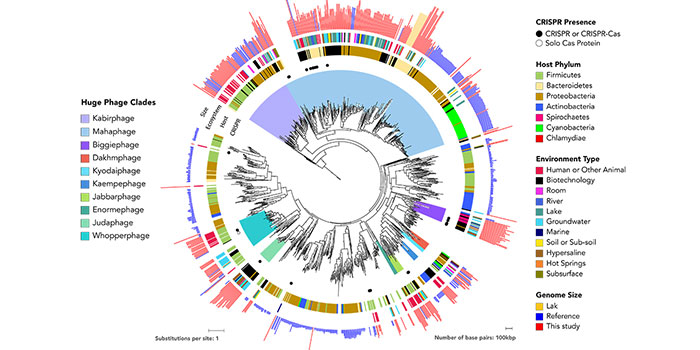
Forskerne har inddelt de mange kæmpe-bakteriofager i grupperinger ud fra deres slægtskab til hinanden. Disse grupper har fået navne, der afspejler forskernes ophav: De slutter alle med det engelske ord for fag (phage), mens den første del er ordet ’kæmpe’ på forskernes modersmål. En af dem hedder således ”kaempephage”.
Læs mere
Forskernes opdagelser er beskrevet i en artikel i det ansete, videnskabelige tidsskrift Nature: Clades of huge phage from across Earth’s ecosystems.
Forskningsgruppen for Genetisk Epidemiologi ved DTU Fødevareinstituttet udfører målrettet forskning med henblik på at forudse og forhindre smitsomme sygdomme blandt mennesker og dyr samt understøtte global detektion og kontrol med særlig fokus på antibiotikaresistens.
Figuren er et slægtstræ over kæmpefager, som forskere har rekonstrueret ved hjælp af viden om fagernes arvemasse. De indre til ydre ringe viser, hvorvidt fagerne indeholder et CRISPR-Cas-system, bakterie-værtens taksonomiske række, i hvilken type prøve, fagerne er fundet og størrelsen på deres genom. De ti nyligt identificerede grupper af kæmpefager er anført til venstre.